生命科学としても知られる生物学は、知識の中核分野の 1 つです。それは生物の重要なプロセスを扱います。この分野の研究開発の歴史は非常に古い。コンピューター技術の発展により、男性はこの分野で真の進歩を遂げました。致命的な病気の克服から生物の謎の解明まで、コンピューターは生物学者にとって素晴らしいパートナーです。オープンソースの生物学ツールは数多くあります。 Linux は、多くの研究者に好まれている非常にカスタマイズ可能なオープン ソース オペレーティング システムです。したがって、Linux 生物学ソフトウェアを探している生物学者またはアマチュアの生物学愛好家であれば、これらの Linux PC 用の生物学ツールをチェックして、研究や研究を最大限に活用することをお勧めします。
Linux に最適な生物学ツール
一部の人々は、Linux には巨大なソフトウェア ライブラリがないという共通の誤解を持っています。しかし、教育および研究ベースのソフトウェアのカテゴリーでは、Linux が依然として無敵であることに驚かれることでしょう。それは、ほとんどの科学者や研究者がオープンソース ソフトウェアの動きに賛同しているからです。
したがって、Linux 用の生物学ツールの広範なコレクションを入手できます。それらは無料で、有料のソフトウェアと同じです。ここでは、探すのに手間がかからないように、さまざまな種類の 15 個のツールの精選されたリストを作成しました。この記事全体を読むことで、あなたのニーズに合った Linux システムに最適なソフトウェアが見つかることを願っています.
1.エンボス
ソフトウェアの名前の説明は、European Molecular Biology Open Software Suite です。これは、生物学の分野に関心のある人向けに作成された Linux 用のオープンソースの生物学ツールです。 EMBOSS は強力なシーケンシャル分析ツールです。このツールの機能と可能性は説明のつかないツールの完全なパッケージです。
EMBOSS の主な機能
- Web から順次データを迅速にクロールして取得できます。
- EMBOSS は、配列アラインメント、タンパク質モチーフの同定、塩基配列パターン解析などに使用されます。
- 新しいオープンソース ツールをリリースするためのライブラリが組み込まれています。
- 取得したデータをすばやく公開するための高度なプレゼンテーション ツールが組み込まれています。
- 追加のプログラミング ライブラリを使用して、文字列処理、パターン マッチング、リスト処理、データベース インデックス作成を実行できます。
- 統合機能は、他の一般的なツールとの同期に役立ちます。
2. NAMD
NAMD は、巨大な生体分子システムをシミュレートするために特別に開発されたシミュレーション プログラムです。この Linux 用の生物学ツールは非常に強力で、一度に何百万もの原子を並列処理できます。 Charm++ は、このプログラムの作成に使用される C++ ベースの言語です。 NAMD は、Converse という名前のランタイム環境を使用して、一度に大量の生物学的データを処理するのに役立つ並列クラスターベースのシステムで実行します。

NAMD の主な機能
- 分子構造シミュレーションは、Visual Molecular Dynamics を使用して作成されます。
- X-PLOR、CHARMM、AMBER など、さまざまな種類の入力ファイルをサポートしています。
- NAMD は数値解析にマルチタイム ステップ積分を使用します。
- ユーザーは幅広いダイナミクス シミュレーション オプションから選択できます。
- GPU アクセラレーション処理をサポートしています。
- このツールは、集合変数モジュールを介してレプリカベースのアンブレラ サンプリングをサポートします。
3. GROMACS
GROMACS は、もう 1 つの生物学的シミュレーション ツールであるだけではありません。むしろ、統合された構築ツールと分析ツールを備えた完全なソフトウェア パッケージです。この多用途の Linux 用生物学ツールは、数千から数百万の生物学的粒子の分析とシミュレーションを実行できます。主に、タンパク質や脂質などの生物学的化学物質の分析用に開発されました。しかし今では、生物学以外の研究分野でも使用されています.
GROMACS の主な機能
- このツールは、競合他社よりも 2 倍から 3 倍高速です。
- ソフトウェア コードは、データ処理を高速化するために高度に最適化されています。
- Gromacs はとても使いやすいです。エラー コードはわかりやすいようにプレーン テキストで記述されています。
- このツールの詳細なユーザー マニュアルは、電子ペーパー形式で無料で入手できます。
- コンパクトな方法で軌跡データを保存できます。
- 軌道分析用の統合ツールがいくつかあります。ユーザーは、この目的のためにコードを記述する必要はありません。
- 非常に便利な、完全に自動化されたタンパク質のトポロジ ビルダーを備えています。
4. VMD
VMD は、Linux 用に開発された高度な生体分子可視化プログラムです。分子可視化プログラムは、主に分子データを3Dグラフィックスで表示するためのプログラムです。 VMD は、PDB または Protein Data Bank ファイルを読み取って分析し、それらを構造化されたグラフィカルな方法でレンダリングできます。さまざまな条件やケースの分子をシミュレートすることもできます。したがって、生物学の深い研究者にとって非常に有用なプログラムになっています.
VMD の主な機能
- コンピュータの外部 GPU 電源を利用できます。
- 開発者は、分子の数やその他のパラメータに制限を適用していません。 RAM が限界です!
- ユーザーは組み込みツールを使用して、標準の 3D 出力から PDF ファイルを簡単に生成できます。
- ステレオ ディスプレイ システムがあれば、VMD を利用できます。
- 組み込みのファイル リーダーの広範なライブラリは、最大 60 の異なるファイル形式をサポートします。
- 研究者は、Tcl 言語を使用してルーチン コマンドを記述できます。
5.シムポップ
SimuPOP は、Linux 用の通常の生物学ツールではありません。むしろ、前向きな集団遺伝シミュレーション環境です。人口に関連する問題を分析およびシミュレートできます。したがって、生物学分野の研究者は、このツールを使用して複雑な病気の蔓延をシミュレートしています。 simuPOP は Python をコア スクリプト言語として使用します。
simuPOP の主な機能
- 集団の個人に情報フィールドを添付するオプションがあります。
- 染色体の相同セットの数またはその他のパラメーターの数に制限があります。
- 母集団分析用の 70 を超える組み込み演算子があります。
- 高度なスクリプト インターフェースにより、ユーザーはこのプログラムをカスタマイズできます。
- simuPOP には、初心者向けの包括的なドキュメント システムがあります。
6.筋肉
MUSCLEは元のソフトウェア名MUSCLE MUの略です 一つ一つ S シーケンス C Lによる比較 og- E 期待。これは、Linux 用の非常に人気のある生物学ツールであり、アミノ酸またはヌクレオチド配列の複数のアラインメントを作成するために使用されます。さらに、その優れた精度と速度により、ClustalW2 や T-Coffee などの他の競合他社よりも優れています。このカテゴリで最速のプログラムの 1 つと考えられています。

マッスルの主な機能
- 3 つの異なるタンパク質プロファイル スコアリング機能をサポートしています。
- MUSCLE は対角線とアンカーの最適化機能を提供します
- このツールでは、一般的なテキストベースの形式である FASTA が入力ファイルと出力ファイルの両方として使用されています。
- LUSTALW、MSF、HTML などのさまざまな一般的な形式で出力ファイルを生成できるという追加の利点があります。
7.シービュー
SeaView は通常の複数配列アラインメント ソフトウェアです。しかし、その特徴は、非常に優れた使いやすいグラフィカル ユーザー インターフェイスを備えていることです。このパッケージは、Clustal Omega、Gblocks、PhyML など、他のさまざまな一般的なツールのバックエンドとして使用されます。一般に FLTK として知られる Fast Light Toolkit は、このプログラムのユーザー インターフェイスを強化します。

SeaView の主な機能
- NEXUS、MSF、CLUSTAL、FASTA、PHYLIP など、DNA およびタンパク質配列決定用のほとんどのファイル形式をサポートしています。
- ユーザーは、アラインメント アルゴリズム用に外部の FASTA 形式のファイルをインポートできます。
- 系統樹を描画し、PDF、SVG、EPS などのさまざまな一般的な形式で生成して、印刷または公開することができます。
- SeaView には、インターネットから遺伝子配列をダウンロードするためのダウンローダーが組み込まれています。
8.ツリーパズル
TREE-PUZZLE はソフトウェア PUZZLE の新しい名前です。これは、Linux 用の非常に人気のある生物学ツールです。もともとは、大規模なデータ セットの分析に使用されるコンソール ベースのツリー検索アルゴリズムです。この TREE-PUZZLE ソフトウェア パッケージは、Strimmer と von Haeseler によって記述されたアルゴリズムを使用してツリーを再構築できます。
TREE-PUZZLE の主な機能
- 四重奏の不可解なアルゴリズムを使用しています。
- このツールは、各内部ブランチのサポートの見積もりを自動的に割り当てることができます。
- TREE-PUZZLE は、ユーザーが指定した木のセットを入力して木を構築できます。
- データセットに対して統計テストを実施するためのツールがいくつかあります。
- パラメータとペアごとの距離を推定できます。
9.ツリービュー X
これは、系統樹を構築するためのオープンソースの生物学ツールです。ツリー構築ソフトウェアは、生物学の分野で非常に重要です。そのため、優れた Linux 生物学ツールと見なされています。さまざまなファイル形式のツリー ファイルを読み取ることができます。
TreeView X の主な機能
- wxWidgets C++ ライブラリに基づく豊富な GUI を備えています。
- さまざまな画像ベースのファイル形式でツリーをエクスポートできます。
- TreeView X には高度な印刷オプションが組み込まれており、ユーザーのニーズに応じて用紙番号をフォーマットするのに役立ちます。
- ドラッグ アンド ドロップ機能により、このツールを使用する際の生産性が向上します。
10.うげね
Linux 用のオープンソースの生物学ソフトウェアです。 UGENEは、さまざまな生体データの解析に利用されています。現在では、主にゲノム配列決定に使用されています。分析されたデータは、コンピュータ ストレージまたは共有ラボ データベースに保存できます。このツールのグラフィカル ユーザー インターフェイスは、ユーザーが事前のコーディングの知識がなくてもこれを操作するのに役立ちます。 GUI 以外にも、従来のコマンドライン インターフェースを使用できます。
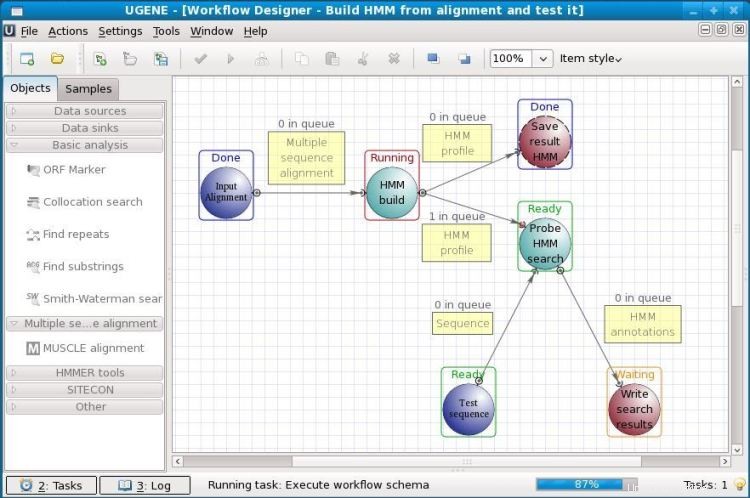
UGENE の主な機能
- ユーザーはタンパク質配列を簡単に作成して注釈を付けることができます。
- ホスト CPU の複数のコアを利用でき、個別のグラフィックス カードを利用できます。
- PDB、NCBI などの一般的なバイオインフォマティクス サーバーとの統合が組み込まれています。
- UGENE には、PCR プライマーを設計するための統合された Primer3 ツールがあります。
- 高度なクロマトグラム ビューアを備えています。
- このツールは、ExpertDiscovery で複雑なシグナルを検索できます。
11.プライマー3
Primer3 は、Linux 用の最も人気のある生物学ソフトウェアの 1 つです。これは、GNU ライセンスに基づく、Linux 用の無料でオープンソースの生物学ツールです。このツールは、DNA 配列からプライマーを選択するために使用されます。このツールには、ローカルにインストールしたくない人のために、Primer3 Plus という名前の代替 Web ユーザー インターフェイスもあります。
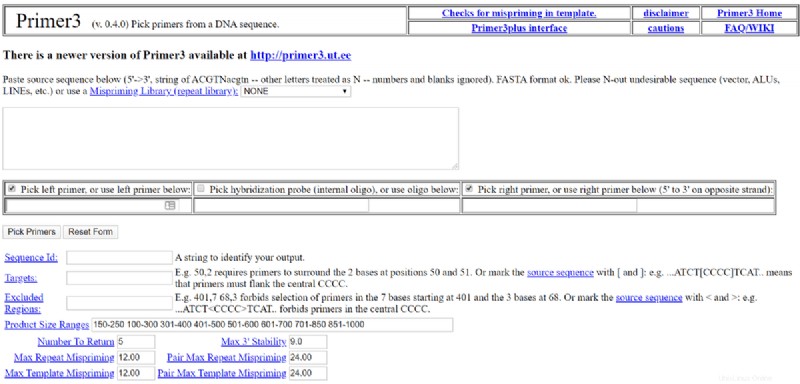
Primer3 の主な機能
- ユーザーは、ほぼすべての一般的なファイル形式でシーケンス ファイルをインポート/アップロードできます。
- シーケンスはプレーン テキストで貼り付けることができます。
- 一般設定と詳細設定のカテゴリに多くのカスタマイズ機能があります。
- ユーザーはこのツールでシーケンスの品質を入力できます。
- このツールには、ペナルティ ウェイト専用のタブがあります。
12.統合されたゲノム ブラウザ
その名の通り、デスクトップ用のゲノムブラウザです。無料でオープンソースの生物学ツールです。この Linux 用生物学ソフトウェアは、インターネットからゲノム配列を検索できます。もちろん、これらの特定のバイオインフォマティクス データは、通常のブラウザから検索できます。しかし、私を信じてください、この専用ブラウザはワークフローを大幅に高速化します.このツールは、Java ライブラリである Genoviz SDK に基づいて構築されています。
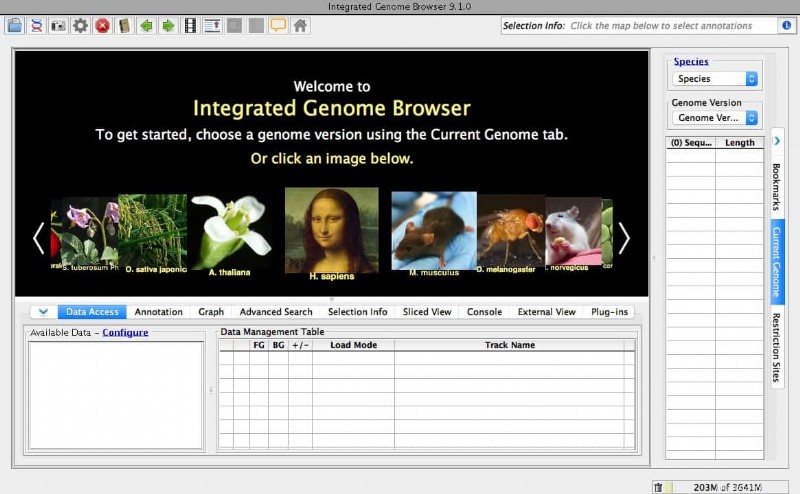
統合されたゲノム ブラウザの主な機能
- このツールは、BAM、BED、Affymetrix CHP、FASTA、GTF、PSL など、さまざまなファイル形式からデータを読み取ることができます。
- ユーザーは出力を SVG、PNG、さらには使いやすい PDF などの印刷可能な形式にエクスポートできます。
- 動的でリアルタイムのズームおよびスクロール機能
- アノテーション機能用の REST スタイルの Web サービスをサポートしています。
13.ランプ
LAMMPS は、最も人気のあるオープンソースの生物学ツールの 1 つです。略語は「L」の略です 巨大スケール A tomic/M オレキュラー M 積極的に P アラレル S エミュレータ。」汎用の分子動力学ソフトウェアです。しかし、今日では、生物学研究の分野で非常に使用されています。これは、サンディア国立研究所によって開発および維持されています。この Linux 生物学ソフトウェアは、研究者間の並行通信に Message Passing Interface または MPI プロトコルを使用します。
LAMMPS の主な機能
- Verlet List という名前の効率的なデータ構造を使用して、近くの粒子を追跡します。
- シミュレーション ドメインをより小さなサブドメインに分割し、プロセッサごとに分散することで、並列コンピューティング システムの可能性を最大限に活用できます。
- このツールは C++ で作成されているため、移植性が高いです。
- CUDA および OpenCL GPU レンダリング システムの組み込みサポート
- ユーザーは新しい機能を簡単に拡張できます。
14.母
Mothur は、学者の間でよく知られている Linux 生物学ソフトウェアです。このソフトウェア プロジェクトは、Patrick Schloss 博士らによって開始されました。これまでのところ、生物学研究の多くの出版物がこのソフトウェアを引用しています。このオープンソース ツールは、非常に効率的なバイオインフォマティクス データ プロセッサです。主に未培養の微生物の DNA 分析に使用されます。
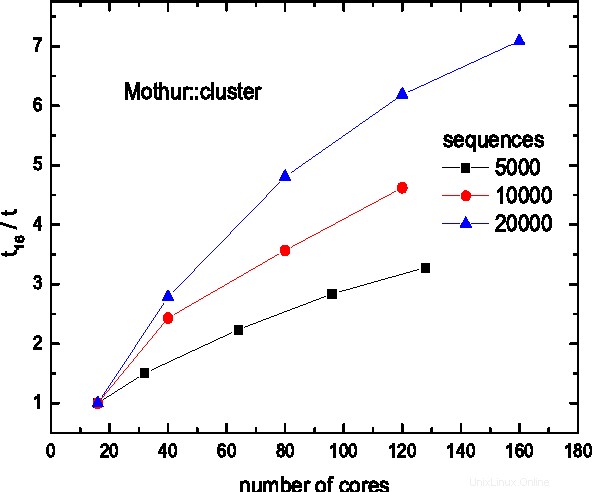
Mothur の主な機能
- 複数の DNA 配列決定法から生成されたデータを処理できる
- 454 パイロシーケンシング、イルミナ HiSeq および MiSeq、Sanger、PacBio、IonTorrent など、一般的な方法のほぼすべてがこのツールでサポートされています。
- 16S rRNA 遺伝子配列の分析において、Mothur に勝るツールは他にありません。
- 著名な生物学者グループによって定期的に管理されています。
15.パスビジオ
PathVisio は、Linux 用の無料でオープンソースの生物学ツールです。生物学的経路の描画、編集、および分析に使用されます。パッケージには多くの便利な機能が組み込まれています。ユーザーは、プラグインを介して追加機能をインストールすることもできます。このツールは Java に基づいているため、Linux を含むあらゆるプラットフォームに簡単にインストールできます。
PathVisio の主な機能
- パスウェイ用の高度な描画ツールと注釈ツール
- さまざまな種類の生物学的経路を分析することさえできます。
- PathVisio には WikiPathways との統合が組み込まれており、簡単に公開できます。
- オープンソース ツールの Cytoscape は、このツールと簡単に統合できます。
- PathVisioRPC を介して他のプログラミング言語と統合できます。
最終的な考え
ご覧のとおり、生物学の分野で必要とされるさまざまな目的のための多数のツールがあります。生物学は、知識と研究の広大な分野です。したがって、上記のすべてのツールを使用する必要がないことは明らかです。この精選された Linux 生物学ソフトウェアのリストを試してみると、どれが自分の作品に最も適しているかがわかります。また、このカテゴリでお気に入りのソフトウェアがあれば、下にコメントして他の人に知らせることができます。