この分野で長い間広く使用されてきた、さまざまな Linux バイオインフォマティクス ツールが利用可能です。バイオインフォマティクスは、多くの点で特徴付けられています。ただし、生物学的情報を分析するための数学、計算、および統計の組み合わせとして定義されることがよくあります。バイオインフォマティクス ツールの主な目標は、効率的なアルゴリズムを開発して、それに応じて配列の類似性を測定できるようにすることです。
Linux に最適なバイオインフォマティクス ツール
この記事は、Linux プラットフォームで利用できるバイオインフォマティクス ツールに焦点を当てて書かれています。すべての効率的なツールについて詳しく説明し、レビューしました。さらに、この記事には重要な機能、プロパティ、およびダウンロード リンクがあります。それでは、見ていきましょう。
1. geワークベンチ
geWorkbench はゲノムで精緻化できます workbench は、統合ゲノミクスで機能する Java ベースのバイオインフォマティクス ツールです。そのコンポーネント アーキテクチャは、複雑なバイオインフォマティクス アプリケーションに構成される特別に開発されたプラグインを容易にします。現在、配列データのサポート、視覚化、分析に使用できるプラグインは 70 以上あります。
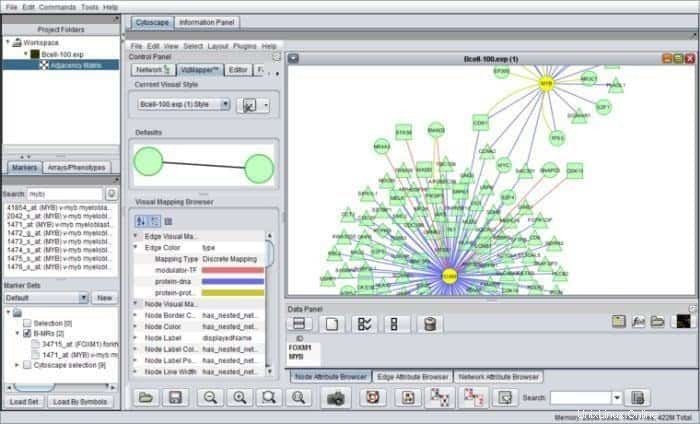
geWorkbench の機能
- t 検定、自己組織化マップ、階層的クラスタリングなど、多くの計算分析ツールに含まれています。
- 分子相互作用ネットワーク、タンパク質構造、タンパク質データが特徴です。
- 遺伝子の統合と注釈の経路を提供し、遺伝子オントロジーの強化分析のために精選されたソースからデータを収集します。
- このツールでは、コンポーネントが入力と出力のプラットフォーム管理と統合されます。
2.バイオパール
BioPerl は、計算分子生物学のバイオインフォマティクス ツールとして Linux プラットフォームで広く使用されている Perl ツールのコレクションです。バイオインフォマティクスの分野で継続的に使用され、標準の CPAN スタイルのセットになっています。この Linux バイオインフォマティクス ツールは十分に文書化されており、Perl モジュールで自由に利用できます。オブジェクト指向であるため、これらのモジュールは相互に依存してタスクを実行します。
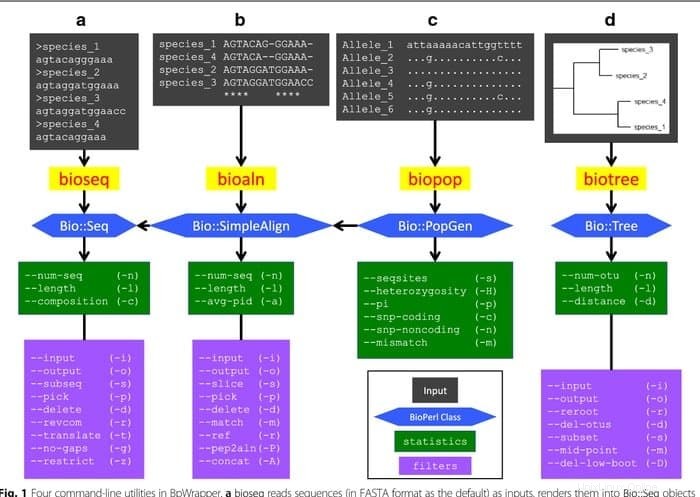
BioPerl の機能
- このバイオインフォマティクス ツールは、ローカルおよび分離されたデータベースから、ヌクレオチドおよびペプチド配列データにアクセスします。
- データベースとファイル レコードの形式を変換するとともに、異なるシーケンスを操作します。
- バイオインフォマティクス検索エンジンとして機能し、ゲノム DNA 上の類似の配列、遺伝子、その他の構造を検索します。
- 配列アラインメントを生成して操作することにより、機械で読み取り可能な配列アノテーションを開発します。
3.うげね
UGENE は無料のオープン ソースであり、Linux 用の統合バイオインフォマティクス ツールのセットです。その共通のユーザー インターフェイスは、ほとんどが使用され、よく知られているバイオインフォマティクス アプリケーションと統合されています。多くの生物学的データ形式は、そのツールキットと互換性があります。したがって、リモート ソースからデータを取得できます。このバイオインフォマティクス ツールは、マルチコア CPU と GPU を利用して最大限のパフォーマンスを提供し、計算活動を最適化します。
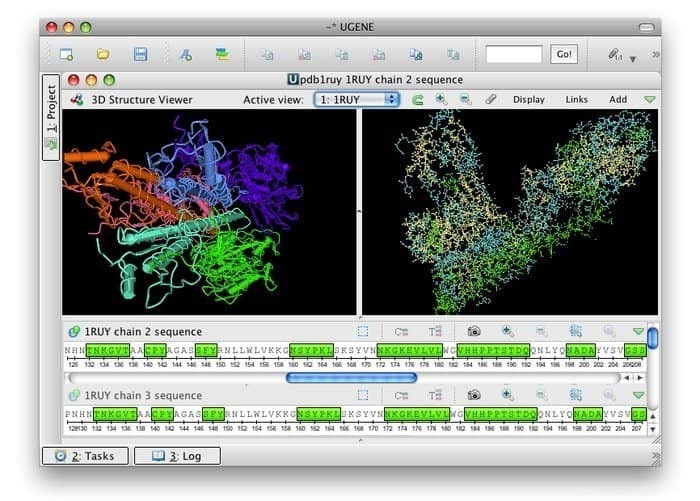
UGENEの特徴
- そのグラフィカル インターフェース ユーザーは、クロマトグラムの視覚化、複数整列エディター、視覚的でインタラクティブなゲノムなど、いくつかの機能を提供します。
- アナグリフ ステレオ モードのサポートとともに、PDB および MMDB 形式の 3D ビューへの道を開きます。
- 系統樹の表示、ドット プロットの視覚化が容易になり、クエリ デザイナーは複雑な注釈パターンを検索できます。
- ワークフロー デザイナー向けのカスタム計算ワークフローへの道を開くことができます。
4.バイオジャバ
Biojava はオープン ソースであり、生物学的データを処理するために必要な Java ツールを提供するプロジェクト専用に設計されています。たとえば、分析および統計ルーチン、一般的なファイル形式のパーサーなど、さまざまなデータセットで機能します。さらに、シーケンスと 3 D 構造の操作を容易にします。この Linux 用のバイオインフォマティクス ツールは、生物学的データセットの迅速なアプリケーション開発を促進することを目的としています。
Biojava の特徴
- クラスファイルとオブジェクトを含み、さまざまなデータセットの Java コードを実装するパッケージです。
- Biojava は、さまざまな目的で使用される Dazzel、Bioclips、Bioweka、Genious などのさまざまなプロジェクトで使用できます。
- DAS クライアントおよびサーバーのサポートとともに、ファイル パーサーで機能します。
- GUI の配列分析を行うために使用され、BioSQL および Ensembl データベースにアクセスできます。
5.バイオパイソン
国際的な開発者チームによって開発され、Python プログラムで記述された Biophython バイオインフォマティクス ツールは、生物学的計算に使用されます。 BLAST、Clustalw、FASTA、Genbank など、さまざまなバイオインフォマティクス ファイル形式へのアクセスを提供し、NCBI や Expasy などのオンライン サービスへのアクセスを可能にします。

Biopython の機能
- インタラクティブで統合された性質を持つシーケンスを作成するための Python モジュールが蓄積されています。
- このバイオインフォマティクス ツールは、翻訳、転写、重量計算など、さまざまな順序で実行できます。
- このツールは特別に強化されています。したがって、タンパク質の構造と配列形式が効率的に管理されます。
- この Linux バイオインフォマティクス ツールはアラインメントに使用できます。したがって、置換行列を作成して処理するための標準を確立できます。
6.インターマイン
InterMine は、生物学データを統合および分析するためのデータ ウェアハウスとして機能する、Linux 用のオープンソースのバイオインフォマティクス ツールです。ソフトウェアであるため、ユーザーはデバイスにインストールして、Web ページでデータを利用できるようにすることができます。これは、データを簡単にドリルダウンできる最も動的なデータ テーブルの 1 つと考えられており、データのフィルタリング方法をスムーズにします。レポート ページに移動するための追加の列はどれですか?
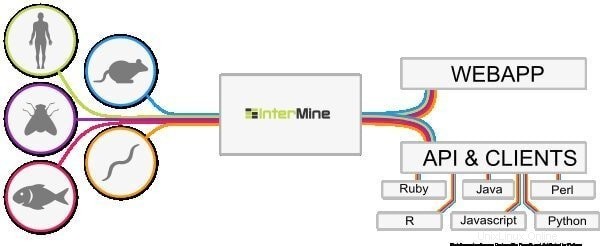
インターマインの特徴
- 遺伝子、タンパク質、結合部位などの単一のオブジェクトと、遺伝子のリストやリスト タンパク質などの複数のリストを処理します。
- 多言語で操作できます。したがって、生体認証情報に関するさまざまなクエリをいくつかの言語で検索できます。
- このソフトウェアでは、テンプレート検索、キーワード検索、クエリ ビルダー、地域検索の 4 つの検索ツールを利用できます。
- Chado、GFF3、FASTA、GO &遺伝子関連ファイル、UniProt XML、PSI XML、In Paranoid オルソログ、Ensembl などのさまざまな形式をサポートしています。
7. IGV
インタラクティブなゲノミクス ビューアとして精巧に作られた IGV は、大規模でインタラクティブなゲノミクス データベースに簡単にアクセスできる最も効果的な視覚化ツールの 1 つと考えられています。配列ベースの次世代シーケンス データに加えて、ゲノム アノテーションを含むさまざまな種類のデータを提供できます。 Google マップと同様に、データセットをナビゲートし、ゲノム全体でシームレスにズームとパンをスムーズに行うことができます。
IGVの特徴
- アラインメント シーケンス リード、突然変異、コピー数など、広範囲のゲノム データセットを柔軟に統合できます。
- 効率的でマルチ解像度のファイル形式を使用することで、大規模なサポート データセットに関するリアルタイムの調査を迅速に行うことができます。
- 数百、場合によっては数千のサンプルの中から、さまざまなデータ タイプを同時に視覚化できます。
- クラウド データ ソースを含むローカルおよびリモート ソースからデータセットを読み込んで、公開されている独自のゲノム データセットを観察できます。
8. GROMACS
GROMACS は、解析および構築ツールに含まれる動的分子シミュレーターです。汎用性のあるパッケージであり、分子動力学に取り組むことを目的としています。たとえば、数百から数千の粒子のニュートン運動方程式をシミュレートできます。複雑な相互作用で結合したタンパク質や脂質など、生化学的分子の初期段階で機能するようにプログラムされています。
GROMACSの特徴
- この Linux インフォマティクス ツールは使いやすく、トポロジとパラメータ ファイルが含まれており、平文で記述されています。
- スクリプト言語は使用されていません。したがって、すべてのプログラムは、入力ファイルと出力ファイル用のシンプルなインターフェイス コマンドライン オプションで操作されます。
- 何か問題が発生した場合、多くのエラー メッセージと整合性チェックが行われます。
- すべてのプログラムは、統合されたグラフィカル ユーザー インターフェースで簡単に操作できます。
9.タベルナ ワークベンチ
Taverna Workbench は、myGrid プロジェクトによって作成されたバイオインフォマティクス ワークフローを設計および実行するようにプログラムされたオープン ソース ツールです。 SOAP や REST Web サービスなど、さまざまなソフトウェアをこのツールに統合できます。欧州バイオインフォマティクス研究所、日本の DNA データバンク、国立バイオテクノロジー情報センター、SoapLab、BioMOBY、EMBOSS などの明確な組織と協力しています。
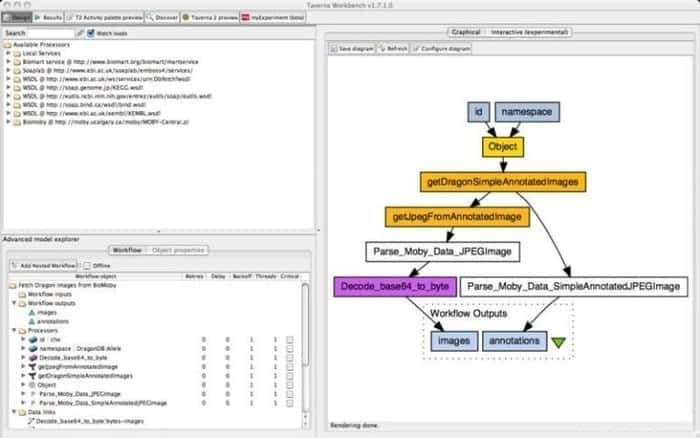
Taverna Workbench の機能
- ワークフローの検索、開発、実行をグラフィカルなワークフローで完全に設計されています。
- 完全にグラフィカルなワークフローで設計されています。さらに、個別のタブがデザインに使用されます。
- 組み込みのヘルプ機能を使用して、ワークフロー、サービス、入力、および出力を説明するための注釈が提供されます。
- ファイルで使用された入力ワークフローを保存できる場合でも、以前に使用されたワークフローはこのツールに保存されます。
10.エンボス
ヨーロッパの分子生物学オープン ソフトウェア スイートを意味する EMBOSS。これは、分子生物学コミュニティのニーズに合わせて開発されたソフトウェアのパッケージです。この Linux バイオインフォマティクス ツールは、さまざまな目的に使用できます。たとえば、さまざまな形式のデータで自動的に機能します。また、Web ページから順次データを収集することもできます。
EMBOSSの特徴
- EMBOSS には、配列アラインメントや配列パターンによる迅速なデータベース検索など、何百ものアプリケーションが含まれています。
- さらに、ドメイン分析やヌクレオチド配列パターン分析など、タンパク質モチーフの同定も行います。
- そのツールキットは、バイオインフォマティクスのアプリケーションとワークフローに対応するように適切に設計されています。
- 他の多くの関連する問題も処理するために、追加のライブラリでプログラムされています。
11.クラスタルオメガ
Clustal Omega はタンパク質に作用し、RNA/DNA は汎用目的で設計された複数配列アラインメント プログラムです。何百万ものデータセットを合理的な時間で効率的に処理できます。さらに、高品質の MSA を生成します。この Linux バイオインフォマティクス ツールには、ユーザーがファイル シーケンスをデフォルト モードのままにしておく必要があるプロセスがあります。それが整列およびクラスター化されてガイド ツリーが生成され、最終的に漸進的な整列シーケンスの形成が可能になります。
クラスタル オメガの特徴
- 既存のアラインメントを相互にアラインメントし、さらに、隠れマルコフ モデルを使用するためにシーケンスをアラインメントにアラインメントすることを容易にします。
- 隠れマルコフ モデルの相同性の新しいシーケンスを参照する、外部プロファイル アラインメントと呼ばれる機能があります。
- Clustal Omega では、Johannes Soeding の HHalign パッケージから取得したアライメント エンジン用の HMM が使用されています。
- Clustal Omega では、プロファイル、配列の整列、HMM の 3 種類の配列入力が可能です。
12.ブラスト
Basic Local Alignment Search Tool または BLAST は、生物学的配列間の類似性を見つけるために使用されます。ヌクレオチド配列とタンパク質配列の間の関連する一致を見つけ、その統計的重要性を示すことができます。クエリ シーケンスは、さまざまな種類の BLAST で構成されています。さらに、このツールは、さまざまな動物の未知の遺伝子で広く栽培されており、定性分析を通じて配列ベースのデータセットをマッピングできます。

BLASTの特徴
- megaBLAST ヌクレオチド - ヌクレオチドは、非常に類似したタイプの配列の検索と最適化を提供します。
- さらに、BLASTN ヌクレオチド - ヌクレオチドは、距離配列を探すため、少し異なる方法で機能します。
- さらに、BLASTP はタンパク質間の関係の発見と比較を実行し、その式は他のさまざまな研究に使用されます。
- TBLASTN は、タンパク質データセットに対するヌクレオチド クエリに焦点を当てており、データベースをその場で翻訳できます。
13.ベッドツール
Bedtool バイオインフォマティクス ソフトウェアは、さまざまなゲノム解析に使用されるスイス アーミー ナイフのようなツールです。ゲノム算術はこのツールを非常に広く使用しており、集合論を見つけることができることを意味します。たとえば、bedtools は、交差のカウント、補完、シャッフル、複数のファイルからのゲノム間隔のマージ、BAM、BED、GFF/GTF、VCF などの特定のゲノム形式の生成を容易にします。
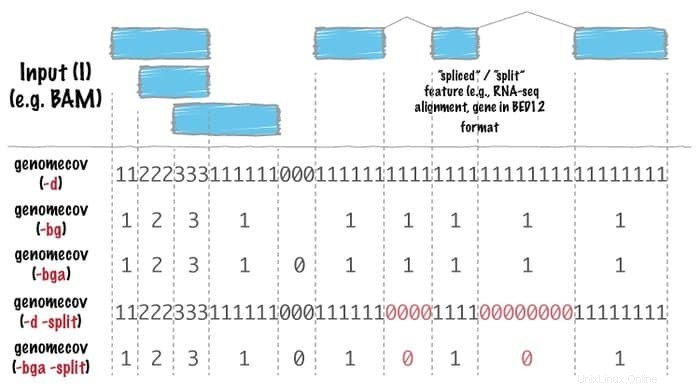
ベッドツールの特徴
- この Linux バイオインフォマティクス ツールでは、それぞれが特に単純なタスクを実行するように設計されています。たとえば、2 つのインターバル ファイルを交差させます。
- ベッドツールを組み合わせて使用することで、複雑で高度な分析が行われます。
- このツールは、グループ研究者によってユタ大学のクインラン研究所で開発されました。
- このツールには多くのオプションがあるため、バイオインフォマティクス分野で多目的に使用できます。
14.バイオクリプス
ライフ サイエンス用のワークベンチで定義されている Bioclipse Linux バイオインフォマティクス ツールは、Java ベースのオープン ソース ソフトウェアです。これは、化学およびバイオインフォマティクスの Eclipse リッチ クライアント プラットフォームを含むビジュアル プラットフォームで動作します。プラグイン アーキテクチャが特徴です。これは、最先端のプラグイン アーキテクチャに加えて、ヘルプ システムなどの Eclipse の機能とビジュアル インターフェイス、ソフトウェアの更新も含まれていることを意味します。
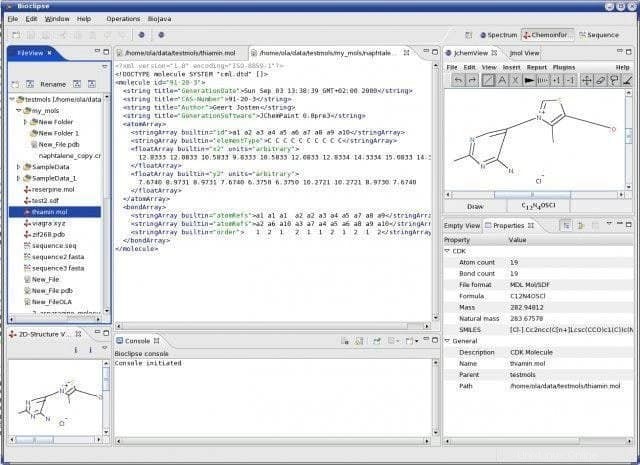
バイオクリプスの特徴
- RNA、DNA、タンパク質などの生物学的配列は、バイオクリプスで管理されます。
- Biojava は、中核となるバイオインフォマティクス機能の提供も支援します。配列アラインメント用のグラフィカル エディターも備えています。
- 代謝の発見部位とともに、薬理学および創薬に使用されます。
- 最後に、セマンティック Web 機能、広範な化合物コレクションの閲覧、化学構造の編集に取り組みます。
15.生体伝導体
Linux プラットフォームで広く使用されているバイオインフォマティクスは、オープンソースで無料のバイオインフォマティクス ツールであり、ハイスループット分析のために医学生物学で首尾一貫して使用されています。主に統計 R プログラミングを使用します。ただし、別のプログラミング言語も含まれています。このソフトウェアは、いくつかの目的に焦点を当てて設計されています。たとえば、共同開発を確立し、革新的なソフトウェアを確実に使用することを目指しています。
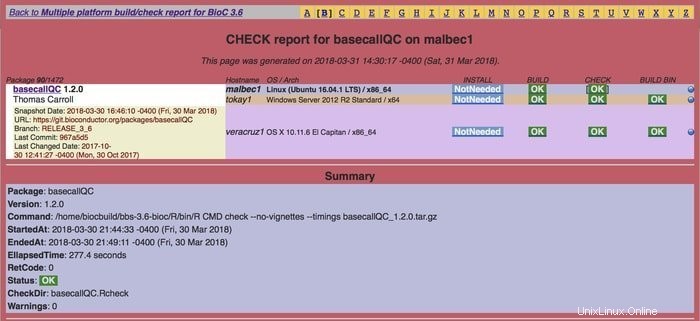
生体伝導体の特徴
- このソフトウェアは、オリゴヌクレオチド アレイ、配列分析、フローサイトメーターなど、さまざまなデータを分析でき、堅牢なグラフィカルおよび統計データベースを生成できます。
- 各パッケージと Binocular パッケージにビネットとドキュメントを含めることで、そのパッケージの機能をタスク指向のテキストで説明できます。
- 関連するマイクロアレイに関するリアルタイム データやその他のゲノム データを、生物学的メタデータとともに生成できます。
- さらに、LIMMA、cDNA Arrays、Affy Arrays、RankProd、SAM、R/maanova、Digital Gene Expression などの発現遺伝子を分析できます。
16.アンフォラ
Automated Phylogenomic infeRence Application の略である AMPHORA は、オープンソースのバイオインフォマティクス ワークフロー ツールです。 AMPHORA2 と呼ばれる別のバージョンの AMPHORA には、細菌と 104 の古細菌の系統発生マーカー遺伝子があります。さらに重要なことに、系統発生データセットと出会った遺伝子データセットの間の情報を作成するために機能します。
アンフォラの特徴
- 単一遺伝子であるため、AMPHORA2 は細菌の分類学的構成を推定するのに最も適しています。
- さらに、メタゲノム ショットガン シーケンスから古細菌群集の分類学的構成を推測することもできます。
- 当初、AMPHORA はサルガッソ海のメタゲノム データの分析に使用されていました。
- しかし、最近では、この点に関して、関連するメタゲノム データを分析するために AMPHORA2 がますます使用されています。
17.アンドゥリル
Anduril は、科学データ分析に関するワークフロー フレームワークを作成するために機能する Linux 用のオープン ソース コンポーネント ベースのバイオインフォマティクス ソフトウェアです。このツールは、ヘルシンキ大学のシステム生物学研究所によって開発されました。この Linux 用のバイオインフォマティクス ツールは、特に生物医学研究分野において、効率的で柔軟かつ体系的なデータ分析を可能にするように設計されています。
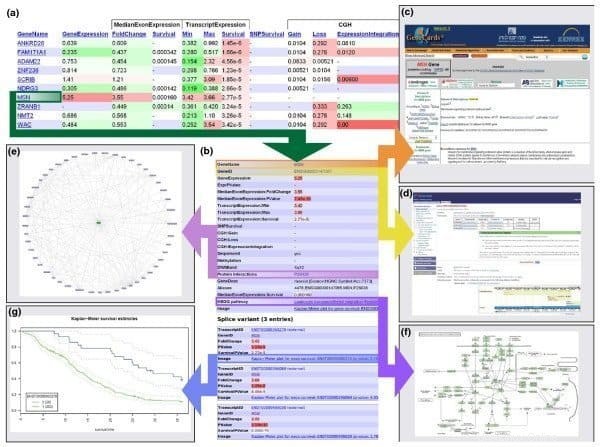
アブドゥリルの特徴
- 異なる処理システムが相互に関連するワークフローで機能します。例えば;プロセスのアウトプットは、他のプロセスのインプットとして機能します。
- 主要な Anduril ツールは Java で記述されていますが、他のコンポーネントは別のアプリケーションで記述されています。
- さまざまな段階で、次のような多数の活動が行われます。データの作成、レポートの生成、データのインポートも行います。
- そのワークフロー構成は、シンプルで明白で強力なスクリプト言語、つまり Andurilscript で行うことができます。
18. LabKey サーバー
LabKey Server は、研究室で研究を統合し、生物医学データを分析および共有するために使用される科学者にとって好ましい選択肢です。このツールでは安全なデータ リポジトリが使用されており、Web ベースのクエリ、レポート、および広範囲のデータベース内でのコラボレーションが容易になります。所定の基礎となるプラットフォームに加えて、このアプリケーションにはさらに多くの科学機器を追加できます。
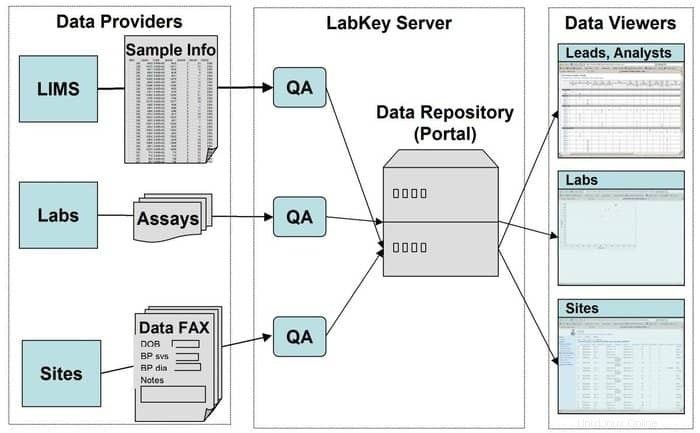
LabKey サーバーの機能
- LabKey サーバーには、あらゆる種類の生物医学データが含まれています。たとえば、フローサイトメトリー、マイクロアレイ、質量分析、マイクロプレート、 ELISpot、ELISA など
- このツールでは、カスタマイズ可能なデータ処理パイプラインが関連するすべてのアクティビティを実行します。
- 参加者の縦断的で大規模な研究の管理をサポートする観察研究が特徴です。
- プロテオミクスは、X! という特定のツールを使用してハイスループット質量分析データを処理するために使用されます。タンデム。
19.母
Mothur は、生物学的データを処理するために生物医学分野で広く使用されているオープンソースのバイオインフォマティクス ツールです。これは、未培養の微生物からの DNA を分析するために頻繁に使用されるソフトウェア パッケージです。 Mothur は Linux のバイオインフォマティクス ツールで、454 パイロシーケンシングを含む DNA シーケンス法から生成されたデータを処理できます。
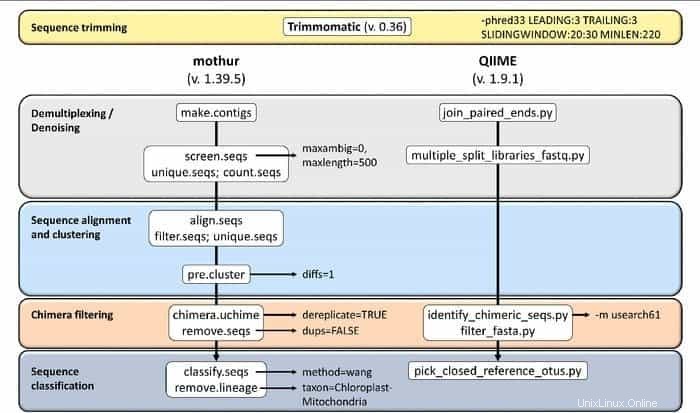
Mothur の特徴
- コミュニティ データの分析とシーケンスの作成を単一のパッケージ ソフトウェアで処理できます。
- このツールでは、大規模なコミュニティ ドキュメント サポートと別の形式のサポートが提供されます。
- Mothur は、16S rRNA 遺伝子配列を分析する最も優れたバイオインフォマティクス ツールであると考えられています。
- このツールには、Sanger、PacBio、IonTorrent、454、および Illumina (MiSeq/HiSeq) の使用方法を知らせる専用のコミュニティとチュートリアルがあります。
20.ボッカ
VOTCA は Versatile Object-directional Toolkit for Coarse-graining Applications の略で、主に分子生物学的データを分析する粗粒度モデリング パッケージを備えた効率的なバイオインフォマティクス ツールとしてブランド化されています。体系的な粗視化技術を開発するとともに、微視的な電荷をシミュレートして無秩序な半導体を輸送することを目的としています。
VOTCAの特徴
- VOTCA は主に、粗視化ツールキット、電荷輸送ツールキット、励起輸送ツールキットの 3 つの主要部分で構成されています。
- 3 つのコア機能はすべて、共有手順を実装する VOTCA ツール ライブラリからのものです。
- VOTCA は、粗視化手法を使用して、関連する活動から最良の結果を導き出します。
- このソフトウェアには、orca DFT パッケージがかなりの範囲でサポートされる励起トランスポート ツールキットが搭載されています。
最終的な考え
全体をカプセル化するために、ここで言及する価値があるのは、4 番目に挙げたバイオインフォマティクス アプリケーションはすべて、この分野で広く使用されているということです。これらの Linux バイオインフォマティクス ツールは、医学、薬理学、創薬、および関連分野で長い間使用されています。最後に、この記事に関して 2 ペニーを残すように求められます。さらに、この記事が価値があると思われる場合は、いいね、共有、コメントを忘れないでください。貴重なコメントをお待ちしております。